TP : Localisation d’une
mutation par électrophorèse sur gel d’agarose
Objectifs :
F Réaliser
une électrophorèse pour séparer les fragments d’ADN d’un phage Lambda muté et
d’un phage Lambda non muté
(bactériophage qui peut infecter Escherichia
coli), digérés par les enzymes EcoRI et HindIII.
F Exploiter les résultats en s’aidant
d’un logiciel de traitement d’image Mesurim afin
d’identifier la souche mutée.
F Localiser précisément la mutation
dans le génome.
Capacités mobilisés : dextérité dans les gestes
techniques, sens de l’observation, raisonnement logique !
Matériel :


Ø ADN
d’un phage Lambda de souche A pur, mélangé à l’endonucléase
EcoRI, à HindIII et à EcoRI + HindIII
Ø ADN
d’un phage Lambda de souche B pur, mélangé à l’endonucléase
EcoRI, à HindIII et à EcoRI + HindIII
Ø Gel
d’agarose avec 8 puits préparé à l’avance
Ø Solution
de tampon 8.3 : TBE
Ø Solution
de bleu de dépôt qui permet d’alourdir l’ADN
et de suivre l’avancée de la migration (déjà mélangé à l’ADN)
Ø Solution
de bleu pour la révélation des bandes : azure A
Ø Eau
distillée
Ø Béchers,
gants, micropipette + cônes, boite en plastique, cuve à électrophorèse,
générateur, fils
NB : compte tenu de la lenteur
de la migration des fragments d’ADN dans le gel, le TP se déroulera en 3 temps :
1- Mise en route de
l’électrophorèse (
1)
Déposer
minutieusement 12 microlitres de chaque tube dans les puits en
respectant l’ordre indiqué au tableau.
2)
Verser le TBE dans la cuve jusqu’à recouvrir le gel de
quelques mm.
3)
Connecter les fils et allumer le générateur avec un voltage
de 100 volts.
4)
Vérifier le bon fonctionnement du dispositif : des
microbulles doivent apparaître à la cathode et à l’anode.
2- Exploitation du résultat obtenu
sur un gel préparé à l’avance (1h30)
1) Etablir la carte de
restriction du phage Lambda non muté en utilisant le document 1.
2) En déduire la taille
des fragments obtenus à l’issue de la digestion par EcoRI,
HindIII et EcoRI + HindIII
3) Représenter sur le
document 2 le profil de migration attendu sur le gel (positions relatives).
4) Comparer votre schéma
au résultat expérimental (attention les petits fragments sont souvent
invisibles sur le gel) et repérer le phage muté.
NB : pour
faire cette comparaison, vous pouvez travailler sur la photo du gel obtenu qui se trouve dans le dossier SVT de la
classe.
5) Identifier l’enzyme qui
va vous permettre de localiser la mutation en justifiant votre choix.
6) Ouvrir l’image
correspondant à la photo interprétée du gel obtenu dans Mesurim.
7) Repérer les fragments
connus et les nouveaux fragments apparus à cause de la mutation.
8) Utiliser les fragments
connus issus de l’ADN digéré par les deux enzymes pour construire la courbe
étalon : Log (taille du fragment en pb) = f(distance en pixels) en utilisant la fonction tableau du
logiciel.
9) Déterminer la longueur
des nouveaux fragments.
NB :
pour gagner du temps, vous pouvez mesurer le spectre d’absorption sur une
droite passant par toutes les bandes en partant de la bordure du gel.
10) En déduire la position
de la mutation sur la carte de restriction.
3- Révélation
des fragments obtenus pendant la séance (
1) Eteindre le générateur
et le débrancher.
2) Retirer le couvercle et
récupérer le gel pour le glisser délicatement dans la boite en plastique.
3) Rincer le gel à l’eau
distillée.
4) Immerger le gel dans
l’azure A pendant
5) Rincer à nouveau le gel
à l’eau distillée voire avec de l’éthanol si la coloration est trop marquée.
6) Comparer ce gel à celui
sur lequel vous avez travaillé.
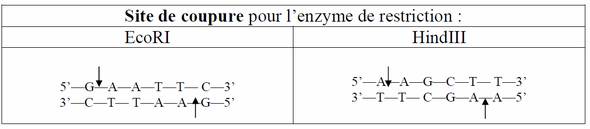
Document 1 : les sites de
coupure des endonucléases EcoRI
et HindIII sur le génome du phage lambda
L’ADN du phage lambda est bicaténaire et constitué de 48502 paires de bases. Son ADN a été entièrement séquencé et l’on connaît les sites de coupures des deux enzymes de restriction EcoR1 et HindIII. Cela permet de disposer de marqueurs de longueurs connues. On peut également, grâce à l’électrophorèse, comprendre l’action des enzymes de restriction en comparant l’action de EcoR1, HindIII et de EcoR1+HindIII sur l’ADN de ce bactériophage.


|
Document
2 : profils de migration attendus à compléter (positions relatives des
fragments) |
|||
|
|
|
|
|
|
ADN de phage lambda |
Digéré par EcoRI |
Digéré par HindIII |
Digéré par EcoRI
et HindIII |