|
TP 13 : le polyallélisme de la globine bêta |
L'hémoglobine est constituée de 2 chaînes polypeptidiques alpha (= globines alpha) et de 2 chaînes polypeptidiques bêta (= globines bêta). On s'intéresse plus particulièrement à la séquence du gène codant pour la globine bêta. Un de ses allèles est très fréquent dans la population (plus de 90% des individus dans la plupart des pays) : cet allèle est considéré comme l'allèle normal (betacod.adn = allèle normal de référence). Les autres allèles sont rares et souvent à l'origine de maladies comme la drépanocytose ou les thalassémies.
Objectifs :
- Comparer plusieurs séquences du gène de la globine bêta à l'aide du logiciel Anagène et identifier les mutations à l'origine des différents allèles. - Comparer les séquences polypeptidiques correspondant aux gènes étudiés et noter les modifications observées. - Mettre en relation les différences constatées au niveau des séquences d'ADN et des séquences polypeptidiques pour comprendre l'origine de la drépanocytose et des thalassémies
I- Protocole à suivre pour comparer les séquences d'ADN
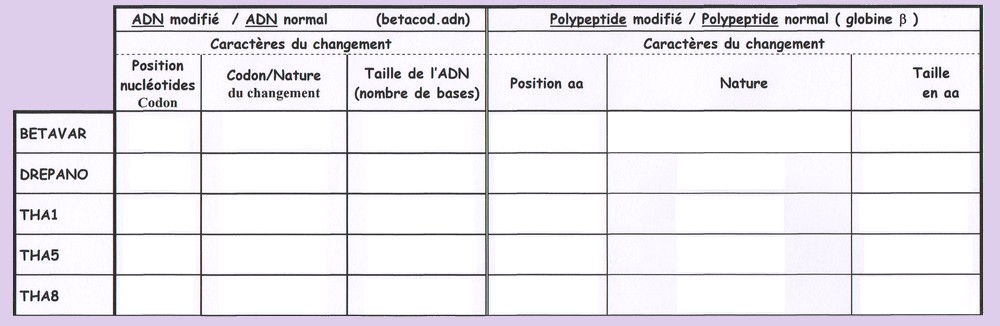
1- Lancez le logiciel Anagène. 2- Chargez les séquences d'ADN en allant dans "fichier", "banque de séquences", "chaînes de l'hémoglobine", "bêta" : Dans "les séquences normales" : "betacod.adn" et "betavar.adn". Dans "les séquences mutées" : dans "drepanocytose", "drepacod.adn" puis dans "thalassémies", "tha1cod.adn", "tha5cod.adn", "tha8cod.adn". 3- Utilisez la fonction "information" (I) pour noter la taille des molécules dans le tableau fourni. 4- Sélectionnez toutes les séquences (betacod.adn doit être en première ligne car c'est l'allèle de référence). 5- Allez dans la barre de menu et choisissez "traiter" puis "comparer les séquences" et "alignement avec discontinuité" (seuls les nucléotides différents sont indiqués) 6- Utilisez la règle et le curseur pour repérer les différences et compléter le tableau.
II- Protocole à suivre pour comparer les séquences protéiques
1- Effacer les comparaisons et les brins d'ADN en cliquant sur la croix en haut à droite des 2 fenêtres. 2- Dans "fichier", "banque de séquences", choisissez "les chaînes de l'hémoglobine" et "bêta" Dans "les séquences normales" : "beta.pro" (betavar.pro n'existe pas : il faudra la créer à partir de betavar.adn en cliquant "traiter", "convertir les séquences", "peptidiques" puis "traduction simple", "résultat dans la fenêtre Affichage/édition". Ce nouveau fichier s'appelle pro-betavar.adn) Dans "les séquences mutées" : dans "drepanocytose", "drep.pro" puis dans "thalassémies", "tha1.pro", "tha5.pro", "tha8.pro". 3- Utilisez la fonction "information" (I) pour noter la taille des molécules dans le tableau fourni. 4- Comparez toutes ces séquences à "beta.pro" (à placer en première ligne) en cliquant "traiter", "comparer les séquences", "comparaison simple" 5- Complétez le tableau

III- Mise en relations des différences constatées au niveau des séquences d'ADN et des séquences polypeptidiques
A partir du tableau rempli et du code génétique (icône "tableau du code génétique"), donnez une explication aux différentes séquences polypeptidiques observées sous forme d'un tableau indiquant pour chaque séquence le type de mutation, l'effet sur la protéine (phénotype moléculaire) et l'effet sur l'individu (phénotype macroscopique).